library(psych) # für deskriptive Statistiken
library(ggplot2) # für Grafiken
library(lavaan) # für den Modelltest
library(semPlot) # zur graphischen Darstellung des ModellsArbeitsauftrag: Konfirmatorische Faktorenanalyse
1 Einführung
In diesem Tutorial werden Sie darauf vorbereitet, wie Sie die Daten aus Ihrem im UK erstellten Fragebogen mithilfe einer Konfirmatorischen Faktorenanalyse (CFA) in R auswerten können. Dazu arbeiten wir zunächst mit einem Beispieldatensatz, an dem wir die grundlegenden Schritte der Auswertung nachvollziehen.
Ein Schwerpunkt wird sein, wie wir Modelltests in R durchführen. Insbesondere schauen wir uns im Detail an, wie man den Modelltest für das eindimensionale \(\tau\)-kongenerische Modell in R durchführt und dabei die Modellparameter schätzt und interpretiert. Zusätzlich zum Modelltest werden wir uns weitere Methoden anschauen, wie man den Modellfit beurteilen kann.
Wir werden diese Methoden zunächst einmal gemeinsam durcharbeiten, bevor Sie den vollständigen Ablauf dann eigenständig im Arbeitsauftrag anwenden.
1.1 Auffrischung der R Grundlagen
Da das Arbeiten mit R bei einigen von Ihnen vielleicht schon eine Weile zurückliegt, lohnt es sich, die wichtigsten Grundschritte noch einmal in unserem R-Tutorial nachzulesen. Dort finden Sie Hinweise zu …
- Abschnitt 2.1: Erstellen und Speichern von Skripten
- Abschnitt 3.1: Laden von Bibliotheken
- Abschnitt 3.2: Auswählen einzelner Spalten
- Abschnitt 2: Anlegen eines RStudio Projekts (Anstelle eines festen Working Directory empfiehlt es sich, ein RStudio Projekt anzulegen, da das oft praktischer ist)
- Abschnitt 3: Datensätze einlesen
1.2 Vorbereitung in R
Bitte führen Sie nun folgende Schritte aus, um unser RStudio Projekt für die Analyse vorzubereiten:
Erstellen Sie ein neues RStudio Projekt (z.B. mit dem Namen Fragebogenentwicklung) und speichern Sie es an einem sinnvollen Ort ab.
Legen Sie ein neues R Skript an (z.B. mit dem Namen Modelltest), und speichern Sie es im Projektordner.
Laden Sie die für die Analyse benötigten Pakete:
Hiweis: Stellen Sie sicher, dass die Pakete vorher installiert wurden.
Laden Sie den Datensatz, den wir in diesem Tutorial verwenden werden, herunter und speichern Sie diesen in Ihrem Projektordner.
Lesen Sie den Datensatz in Ihrem Skript mit dem Befehl
read.csv()ein (wir verwenden hier nichtread.csv2(), da der Datensatz im englischen .csv-Format vorliegt). Weisen Sie dem Datensatz das Objektneozu:neo <- read.csv("NEO_original.csv")
1.3 Kurze Wiederholung: Befehle für Deskriptivstatistik
# Spalte "E1" auswählen
neo$E1
# Spalten "E1" und "E2" auswählen
neo[, c("E1", "E2")]
# Deskriptive Statistiken für die Spalten "E1", "E2", "E3", "E4"
describe(neo[, c("E1", "E2", "E3", "E4")])
# Verteilung des Items "E1"
barplot(table(neo$E1)) # oder
hist(neo$E1) # oder
ggplot(neo, aes(x = E1)) + geom_bar()
# Streudiagramm
plot(neo$E1, neo$E3) # oder
ggplot(neo, aes(x = E1, y = E2)) + geom_jitter()
# Alle paarweisen Streudiagramme
pairs.panels(neo[, c("E1", "E2", "E3", "E4")],
density = FALSE,
jiggle = TRUE,
smooth = FALSE)# Spalte "E1" auswählen
neo$E1 [1] 3 0 4 2 3 0 2 3 3 2 2 3 1 1 2 4 1 1 1 1 4 3 3 2 3 2 2 3 1 1 2 4 3 2 1 3 3
[38] 2 3 2 3 3 2 2 2 2 4 2 3 2 2 4 3 2 1 2 3 3 4 3 2 1 1 1 2 2 1 3 3 2 3 1 3 2
[75] 3 3 2 4 0 4 1 2 3 3 2 3 3 2 2 3 2 1 2 3 3 2 3 2 1 1 2 1 1 3 3 2 1 1 2 2 2
[112] 1 3 2 3 4 1 4 3 3 1 4 3 2 2 1 1 4 3 1 0 4 1 2 2 3 2 1 3 2 4 1 2 2 3 1 1 3
[149] 1 2 3 3 2 2 0 2 1 2 2 0 1 3 1 2 1 3 3 1 4 4 2 1 2 2 3 1 3 2 2 2 3 2 1 2 3
[186] 2 3 2 1 3 3 3 3 2 2 2 3 2 2 3 2 3 3 3 2 3 1 3 3 3 1 4 4 3 1 3 4 2 3 3 2 3
[223] 1 1 2 0 1 3 1 3 3 2 2 2 2 4 3 3 0 2 3 3 3 3 2 2 3 2 2 4 3 3 1 4 2 3 3 2 1
[260] 1 1 2 3 3 4 2 3 2 2 4 2 4 2 2 3 3 3 4 1 2 1 0 2 3 2 1 1 3 3 4 2 3 3 1 3 2
[297] 3 2 2 3 2 3 3 3 2 3 2 1 2 3 4 2 1 3 1 0 3 0 3 1 2 1 2 1 2 3 2 3 3 3 2 3 1
[334] 3 3 1 2 1 4 1 3 1 1 2 3 2 2 1 3 2 2 3 4 2 2 2 2 0 0 2 2 3 2 2 0 3 1 1 1 3
[371] 1 0 3 2 1 1 2 1 2 1 1 2 3 3 2 4 2 1 3 2 2 1 2 3 3 2 2 2 3 1 2 2 2 3 3 2 2
[408] 1 2 3 3 1 1 3 3 3 2 4 0 2 2 4 4 3 1 1 1 1 3 2 1 4 3 1 2 1 3 2 3 1 2 1 1 2
[445] 3 3 3 3 3 3 3 2 2 4 3 3 3 2 1 3 4 3 2 1 3 1 2 3 2 1 2 2 2 2 3 3 3 2 3 1 3
[482] 1 3 1 1 2 2 3 4 1 3 3 2 1 3 2 4 2 0 2 3 1 1 3 2 2 3 2 3 3 2 3 2 2 1 1 2 1
[519] 2 1 2 2 3 2 2 4 3 3 2 2 1 1 2 2 1 2 1 1 4 3 3 4 2 1 0 2 2 3 3 2 2 3 3 1 2
[556] 2 3 2 3 4 4 4 3 1 3 3# Spalten "E1" und "E2" auszuwählen
neo[, c("E1", "E2")] E1 E2
1 3 2
2 0 2
3 4 3
4 2 2
5 3 1
6 0 3
7 2 3
8 3 3
9 3 3
10 2 3
11 2 1
12 3 3
13 1 3
14 1 3
15 2 3
16 4 4
17 1 2
18 1 3
19 1 4
20 1 0
21 4 2
22 3 3
23 3 3
24 2 3
25 3 3
26 2 1
27 2 2
28 3 2
29 1 1
30 1 1
31 2 3
32 4 4
33 3 4
34 2 3
35 1 1
36 3 4
37 3 3
38 2 2
39 3 3
40 2 3
41 3 1
42 3 2
43 2 3
44 2 3
45 2 3
46 2 3
47 4 4
48 2 1
49 3 3
50 2 2
51 2 4
52 4 4
53 3 3
54 2 2
55 1 2
56 2 2
57 3 2
58 3 3
59 4 3
60 3 2
61 2 4
62 1 3
63 1 0
64 1 3
65 2 3
66 2 3
67 1 1
68 3 3
69 3 3
70 2 3
71 3 3
72 1 3
73 3 2
74 2 2
75 3 3
76 3 4
77 2 3
78 4 4
79 0 3
80 4 3
81 1 3
82 2 4
83 3 2
84 3 2
85 2 3
86 3 3
87 3 3
88 2 2
89 2 3
90 3 4
91 2 3
92 1 3
93 2 2
94 3 3
95 3 2
96 2 1
97 3 1
98 2 1
99 1 2
100 1 3
101 2 3
102 1 3
103 1 3
104 3 2
105 3 3
106 2 2
107 1 2
108 1 2
109 2 2
110 2 3
111 2 3
112 1 2
113 3 3
114 2 2
115 3 3
116 4 3
117 1 3
118 4 4
119 3 3
120 3 3
121 1 2
122 4 3
123 3 4
124 2 2
125 2 3
126 1 3
127 1 2
128 4 3
129 3 3
130 1 1
131 0 4
132 4 3
133 1 2
134 2 1
135 2 3
136 3 2
137 2 2
138 1 2
139 3 4
140 2 2
141 4 2
142 1 2
143 2 1
144 2 3
145 3 2
146 1 3
147 1 4
148 3 2
149 1 4
150 2 1
151 3 2
152 3 3
153 2 3
154 2 2
155 0 1
156 2 1
157 1 3
158 2 3
159 2 3
160 0 3
161 1 2
162 3 3
163 1 3
164 2 3
165 1 2
166 3 4
167 3 4
168 1 3
169 4 4
170 4 3
171 2 3
172 1 3
173 2 3
174 2 2
175 3 1
176 1 3
177 3 4
178 2 2
179 2 2
180 2 1
181 3 2
182 2 4
183 1 2
184 2 3
185 3 3
186 2 3
187 3 1
188 2 3
189 1 3
190 3 3
191 3 3
192 3 3
193 3 4
194 2 3
195 2 3
196 2 2
197 3 3
198 2 3
199 2 3
200 3 4
201 2 3
202 3 4
203 3 2
204 3 3
205 2 4
206 3 3
207 1 2
208 3 3
209 3 3
210 3 2
211 1 1
212 4 3
213 4 2
214 3 3
215 1 3
216 3 3
217 4 2
218 2 3
219 3 3
220 3 2
221 2 3
222 3 3
223 1 2
224 1 2
225 2 3
226 0 3
227 1 3
228 3 2
229 1 2
230 3 3
231 3 3
232 2 3
233 2 3
234 2 3
235 2 2
236 4 4
237 3 3
238 3 4
239 0 1
240 2 1
241 3 3
242 3 3
243 3 3
244 3 2
245 2 2
246 2 1
247 3 2
248 2 3
249 2 3
250 4 3
251 3 4
252 3 3
253 1 4
254 4 4
255 2 3
256 3 3
257 3 3
258 2 2
259 1 3
260 1 1
261 1 4
262 2 4
263 3 3
264 3 3
265 4 3
266 2 2
267 3 2
268 2 3
269 2 3
270 4 4
271 2 4
272 4 2
273 2 2
274 2 3
275 3 3
276 3 2
277 3 1
278 4 3
279 1 3
280 2 3
281 1 1
282 0 3
283 2 3
284 3 2
285 2 3
286 1 3
287 1 3
288 3 3
289 3 3
290 4 4
291 2 3
292 3 2
293 3 2
294 1 2
295 3 3
296 2 3
297 3 3
298 2 3
299 2 4
300 3 3
301 2 3
302 3 4
303 3 2
304 3 4
305 2 3
306 3 3
307 2 4
308 1 4
309 2 3
310 3 4
311 4 2
312 2 2
313 1 4
314 3 3
315 1 1
316 0 3
317 3 3
318 0 4
319 3 3
320 1 3
321 2 3
322 1 4
323 2 1
324 1 2
325 2 4
326 3 3
327 2 2
328 3 2
329 3 3
330 3 3
331 2 2
332 3 3
333 1 3
334 3 4
335 3 3
336 1 3
337 2 2
338 1 2
339 4 1
340 1 4
341 3 2
342 1 1
343 1 3
344 2 3
345 3 3
346 2 3
347 2 2
348 1 3
349 3 3
350 2 3
351 2 2
352 3 3
353 4 4
354 2 3
355 2 4
356 2 1
357 2 2
358 0 4
359 0 2
360 2 3
361 2 2
362 3 3
363 2 4
364 2 4
365 0 2
366 3 2
367 1 1
368 1 3
369 1 3
370 3 3
371 1 3
372 0 0
373 3 3
374 2 2
375 1 3
376 1 2
377 2 3
378 1 2
379 2 3
380 1 2
381 1 1
382 2 2
383 3 4
384 3 3
385 2 2
386 4 3
387 2 2
388 1 3
389 3 3
390 2 3
391 2 3
392 1 2
393 2 3
394 3 2
395 3 3
396 2 3
397 2 3
398 2 3
399 3 2
400 1 3
401 2 3
402 2 3
403 2 3
404 3 3
405 3 3
406 2 4
407 2 3
408 1 3
409 2 2
410 3 3
411 3 3
412 1 1
413 1 3
414 3 3
415 3 3
416 3 3
417 2 2
418 4 3
419 0 3
420 2 1
421 2 3
422 4 4
423 4 2
424 3 3
425 1 2
426 1 2
427 1 4
428 1 3
429 3 4
430 2 2
431 1 4
432 4 4
433 3 3
434 1 2
435 2 2
436 1 4
437 3 3
438 2 3
439 3 3
440 1 3
441 2 3
442 1 3
443 1 3
444 2 3
445 3 3
446 3 4
447 3 3
448 3 3
449 3 3
450 3 3
451 3 3
452 2 2
453 2 2
454 4 3
455 3 4
456 3 3
457 3 4
458 2 4
459 1 1
460 3 1
461 4 3
462 3 3
463 2 4
464 1 3
465 3 4
466 1 2
467 2 3
468 3 3
469 2 3
470 1 2
471 2 0
472 2 3
473 2 3
474 2 2
475 3 4
476 3 2
477 3 3
478 2 3
479 3 4
480 1 2
481 3 3
482 1 3
483 3 4
484 1 1
485 1 2
486 2 3
487 2 3
488 3 3
489 4 3
490 1 2
491 3 2
492 3 3
493 2 3
494 1 4
495 3 3
496 2 3
497 4 3
498 2 3
499 0 4
500 2 3
501 3 3
502 1 0
503 1 2
504 3 3
505 2 4
506 2 3
507 3 4
508 2 4
509 3 3
510 3 3
511 2 1
512 3 3
513 2 4
514 2 2
515 1 3
516 1 1
517 2 3
518 1 3
519 2 3
520 1 2
521 2 2
522 2 3
523 3 3
524 2 2
525 2 1
526 4 4
527 3 4
528 3 2
529 2 2
530 2 1
531 1 2
532 1 3
533 2 3
534 2 2
535 1 3
536 2 2
537 1 3
538 1 3
539 4 4
540 3 4
541 3 3
542 4 3
543 2 2
544 1 2
545 0 4
546 2 3
547 2 3
548 3 2
549 3 3
550 2 3
551 2 3
552 3 3
553 3 2
554 1 3
555 2 2
556 2 4
557 3 1
558 2 3
559 3 3
560 4 3
561 4 3
562 4 2
563 3 4
564 1 2
565 3 4
566 3 3# Deskriptive Statistiken für die Spalten "E1", "E2", "E3", "E4"
describe(neo[, c("E1", "E2", "E3", "E4")]) vars n mean sd median trimmed mad min max range skew kurtosis se
E1 1 566 2.21 0.98 2 2.20 1.48 0 4 4 -0.12 -0.60 0.04
E2 2 566 2.72 0.85 3 2.78 0.00 0 4 4 -0.57 0.22 0.04
E3 3 566 2.47 1.09 3 2.50 1.48 0 4 4 -0.33 -0.81 0.05
E4 4 566 2.93 0.84 3 2.99 1.48 0 4 4 -0.69 0.52 0.04# Verteilung des Items "E1"
barplot(table(neo$E1)) # oder 
hist(neo$E1) # oder 
ggplot(neo, aes(x = E1)) + geom_bar()
# Streudiagramm
plot(neo$E1, neo$E3) # oder 
ggplot(neo, aes(x = E1, y = E2)) + geom_jitter()
# Alle paarweisen Streudiagramme
pairs.panels(neo[, c("E1", "E2", "E3", "E4")],
density = FALSE,
jiggle = TRUE,
smooth = FALSE)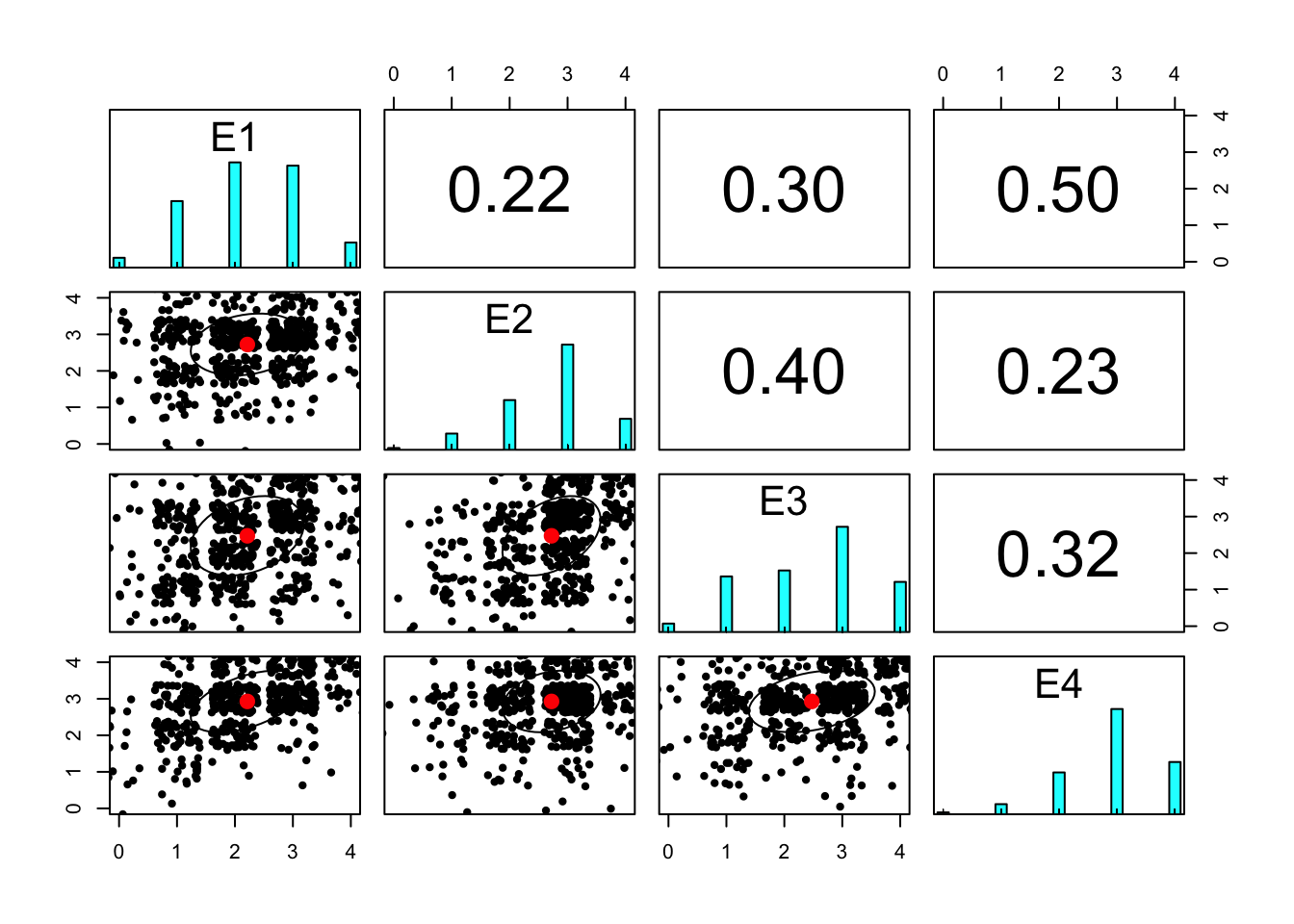
Mit diesen Schritten und Wiederholungen sind wir gut vorbereitet, um uns nun mit der Analyse von Testmodellen zu beschäftigen.
2 Konfirmatorische Faktorenanalyse (CFA)
2.1 Ziel der CFA
In diesem Abschnitt führen wir eine konfirmatorische Faktorenanalyse (CFA) mit dem {lavaan} Paket in R durch. Ziel ist es, das \(\tau\)-kongenerische Modell für den Faktor Extraversion anhand der Items E1, E2, …, E12 zu testen. Das \(\tau\)-kongenerische Modell nimmt an, dass alle Items eine latente Variable (hier: Extraversion) messen, aber unterschiedliche Gewichtungen bzw. Steigungen aufweisen können.
2.2 Schritte der CFA
1. Bestimmung der Anzahl an latenten Variablen
Da wir hier nur die Items E1, E2, …, E12 analysieren wollen, gehen wir für unser Modell davon aus, dass den Items nur eine einzige latente Variable Extraversion zugrunde liegt.
2. Spezifikation des Testmodells
Wir wollen hier ein eindimensionales \(\tau\)-kongenerische Modell betrachten. Zunächst spezifizieren wir das Modell im Textformat und weisen es dem Objekt model zu. Die Beschreibung des Modells folgt der speziellen Syntax des {lavaan} Pakets. Die latente Variable Extraversion nennen wir hier einfach e, und alle Items, die Extraversion messen, werden durch den Ausdruck e =~ E1 + E2 + ... + E12 mit dieser latenten Variable verbunden.
model <- "e =~ E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12" 3. Schätzung des Testmodells
Mit cfa() wird das Modell basierend auf den Daten in neo geschätzt. Die Ergebnisse und Parameterschätzungen werden im Objekt model_fit_extra gespeichert. Das Argument std.lv = TRUE sorgt dafür, dass die Varianz der latenten Variable auf 1 fixiert wird (siehe Normierung beim \(\tau\)-kongenerischen Modell in der Vorlesung). Mit meanstructure = TRUE wird festgelegt, dass Schwierigkeitsparameter für alle Items geschätzt werden sollen.
model_fit_extra <- cfa(model = model, data = neo,
std.lv = TRUE, meanstructure = TRUE)4. Evaluation der Modellpassung
Der folgende Output ist die Grundlage für die Beurteilung der Modellpassung sowie die Interpretation der Modellparameter.
Mit der Funktion summary() wird das Ergebnis des Modelltests und die Parameterschätzungen angezeigt. Mit dem Argument fit.measures = TRUE werden zusätzlich Fit-Indizes und Informationskriterien berechnet. Das Argument standardized = TRUE sorgt dafür, dass auch die Schätzwerte für die standardisierten Modellparameter angezeigt werden.
summary(model_fit_extra, standardized = TRUE, fit.measures = TRUE)lavaan 0.6.17 ended normally after 16 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 36
Number of observations 566
Model Test User Model:
Test statistic 495.835
Degrees of freedom 54
P-value (Chi-square) 0.000
Model Test Baseline Model:
Test statistic 1827.444
Degrees of freedom 66
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 0.749
Tucker-Lewis Index (TLI) 0.693
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -8889.082
Loglikelihood unrestricted model (H1) -8641.165
Akaike (AIC) 17850.164
Bayesian (BIC) 18006.353
Sample-size adjusted Bayesian (SABIC) 17892.070
Root Mean Square Error of Approximation:
RMSEA 0.120
90 Percent confidence interval - lower 0.111
90 Percent confidence interval - upper 0.130
P-value H_0: RMSEA <= 0.050 0.000
P-value H_0: RMSEA >= 0.080 1.000
Standardized Root Mean Square Residual:
SRMR 0.083
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
e =~
E1 0.517 0.041 12.636 0.000 0.517 0.530
E2 0.418 0.036 11.668 0.000 0.418 0.494
E3 0.788 0.042 18.721 0.000 0.788 0.725
E4 0.425 0.036 11.951 0.000 0.425 0.505
E5 0.378 0.046 8.218 0.000 0.378 0.360
E6 0.306 0.046 6.689 0.000 0.306 0.297
E7 0.502 0.042 12.039 0.000 0.502 0.508
E8 0.710 0.033 21.682 0.000 0.710 0.806
E9 0.751 0.043 17.282 0.000 0.751 0.683
E10 0.094 0.048 1.955 0.051 0.094 0.089
E11 0.471 0.040 11.860 0.000 0.471 0.501
E12 0.400 0.048 8.308 0.000 0.400 0.364
Intercepts:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.E1 2.214 0.041 53.964 0.000 2.214 2.268
.E2 2.723 0.036 76.530 0.000 2.723 3.217
.E3 2.473 0.046 54.123 0.000 2.473 2.275
.E4 2.928 0.035 82.641 0.000 2.928 3.474
.E5 1.809 0.044 40.947 0.000 1.809 1.721
.E6 1.776 0.043 40.876 0.000 1.776 1.718
.E7 1.760 0.042 42.342 0.000 1.760 1.780
.E8 2.673 0.037 72.250 0.000 2.673 3.037
.E9 2.337 0.046 50.566 0.000 2.337 2.125
.E10 1.963 0.045 43.909 0.000 1.963 1.846
.E11 2.389 0.039 60.555 0.000 2.389 2.545
.E12 1.890 0.046 40.876 0.000 1.890 1.718
Variances:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.E1 0.685 0.044 15.717 0.000 0.685 0.720
.E2 0.541 0.034 15.907 0.000 0.541 0.756
.E3 0.560 0.041 13.621 0.000 0.560 0.474
.E4 0.529 0.033 15.854 0.000 0.529 0.745
.E5 0.962 0.059 16.402 0.000 0.962 0.870
.E6 0.974 0.059 16.551 0.000 0.974 0.912
.E7 0.725 0.046 15.837 0.000 0.725 0.742
.E8 0.271 0.024 11.488 0.000 0.271 0.350
.E9 0.646 0.045 14.317 0.000 0.646 0.534
.E10 1.122 0.067 16.800 0.000 1.122 0.992
.E11 0.659 0.042 15.871 0.000 0.659 0.749
.E12 1.050 0.064 16.392 0.000 1.050 0.868
e 1.000 1.000 1.000Globaler Modellfit: Modelltest, Fit-Indices und Informationskriterien
Der Block \(\texttt{Model Test User Model}\) im summary-Output liefert den Wert der Teststatistik, die Freiheitsgrade (df) und den p-Wert für den Modelltest. Die Nullhypothese testet, ob die Folgerungen aus dem \(\tau\)-kongenerischen Modell beibehalten werden können. Wenn die Alternativhypothese gilt, bedeutet dies, dass die Folgerungen verletzt sind und das \(\tau\)-kongenerische Modell würde unter der H1 abgelehnt werden. Unsere Daten liefern einen p-Wert von \(p < 0.05\). Wir entscheiden uns also für die H1 und können nicht davon ausgehen, dass das eindimensionale \(\tau\)-kongenerische Modell in der Population gilt.
Die Fit-Indizes CFI, RMSEA, und SRMR zeigen basierend auf den groben Richtwerten nach Hu & Bentler (1998) ein gemischtes Bild: Der RMSEA fällt zu hoch aus (Ziel: \(RMSEA \leq 0.06\) bei \(n > 250\)). Der CFI ist zu niedrig (Ziel: \(CFI \geq 0.95\)). Nur der SRMR ist in Ordnung (Ziel: \(SRMR \leq 0.11\)).
Die Informationskriterien AIC und BIC werden zwar im Output berichtet, sind jedoch ohne ein Vergleichsmodell nicht hilfreich, da die absoluten Werte nicht wirklich interpretierbar sind.
Lokaler Modellfit: Inspektion der geschätzten Parameter und Modifikationsindizes
Der summary-Output enthält auch die Parameterschätzungen für das geprüfte Modell. In den Spalten \(\texttt{z-value}\) und \(\texttt{P(>|z|)}\) finden wir jeweils das Ergebnis der Signifikanztests, deren Nullhypothese jeweils besagt, dass der entsprechende Parameter in der Population den Wert 0 aufweist. Für die allermeisten im Modell geschätzten Parameter entscheidet sich der Test für die Alternativhypothese, dass sich der Wert in der Population von 0 unterscheidet. Für das Item E10 ist jedoch die Ladung nicht signifikant. Damit liegt hier eine mögliche Verletzung des lokalen Modellfits vor. Eventuell lädt das Item nicht auf die latente Variable Extraversion und trägt damit nichts zu deren Messung bei.
Da sowohl der globale als auch der lokale Modellfit Probleme aufzeigen, interessieren wir uns natürlich dafür, welche (Mis-)Spezifikationen des Modells dafür verantwortlich sein könnten. Dafür können wir uns mit der Funktion modindices() die Modifikationsindizes für das Modell ausgeben lassen. Das Argument sort. = TRUE sorgt dafür, dass die höchsten Modifikationsindizes ganz oben angezeigt werden.
modindices(model_fit_extra, sort. = TRUE, standardized = FALSE) lhs op rhs mi epc
41 E1 ~~ E4 65.824 0.220
43 E1 ~~ E6 51.479 0.257
81 E5 ~~ E10 50.790 0.315
42 E1 ~~ E5 40.072 0.227
64 E3 ~~ E8 37.789 0.160
83 E5 ~~ E12 33.339 0.251
89 E6 ~~ E12 30.790 0.241
93 E7 ~~ E11 28.373 0.165
47 E1 ~~ E10 28.137 0.202
95 E8 ~~ E9 25.663 0.133
98 E8 ~~ E12 24.070 -0.137
70 E4 ~~ E6 20.299 0.141
45 E1 ~~ E8 20.229 -0.107
79 E5 ~~ E8 19.694 -0.118
102 E10 ~~ E11 17.908 0.158
55 E2 ~~ E8 16.510 0.085
85 E6 ~~ E8 15.881 -0.106
40 E1 ~~ E3 15.380 -0.121
61 E3 ~~ E5 14.572 -0.134
76 E4 ~~ E12 14.306 0.124
73 E4 ~~ E9 14.104 -0.105
96 E8 ~~ E10 13.326 -0.102
66 E3 ~~ E10 12.907 -0.133
58 E2 ~~ E11 12.632 -0.095
49 E1 ~~ E12 10.820 0.123
69 E4 ~~ E5 10.678 0.102
72 E4 ~~ E8 10.355 -0.067
59 E2 ~~ E12 8.469 -0.096
78 E5 ~~ E7 7.628 0.101
82 E5 ~~ E11 7.062 0.093
46 E1 ~~ E9 6.959 -0.084
57 E2 ~~ E10 6.001 -0.083
67 E3 ~~ E11 5.581 -0.071
74 E4 ~~ E10 5.518 0.078
99 E9 ~~ E10 5.036 -0.087
48 E1 ~~ E11 4.693 0.066
50 E2 ~~ E3 4.423 0.057
52 E2 ~~ E5 4.176 -0.065
60 E3 ~~ E4 4.122 -0.054
90 E7 ~~ E8 4.010 -0.049
86 E6 ~~ E9 3.544 -0.069
80 E5 ~~ E9 3.210 -0.066
68 E3 ~~ E12 3.088 -0.064
39 E1 ~~ E2 2.607 -0.044
103 E10 ~~ E12 2.508 0.073
100 E9 ~~ E11 1.979 -0.044
77 E5 ~~ E6 1.759 -0.055
62 E3 ~~ E6 1.581 -0.044
71 E4 ~~ E7 1.484 -0.034
88 E6 ~~ E11 1.475 -0.043
104 E11 ~~ E12 1.060 0.038
44 E1 ~~ E7 1.032 -0.032
65 E3 ~~ E9 0.900 0.031
92 E7 ~~ E10 0.848 0.036
84 E6 ~~ E7 0.844 -0.034
75 E4 ~~ E11 0.649 0.021
97 E8 ~~ E11 0.627 -0.018
54 E2 ~~ E7 0.480 -0.019
51 E2 ~~ E4 0.463 -0.016
56 E2 ~~ E9 0.304 -0.016
91 E7 ~~ E9 0.270 0.017
63 E3 ~~ E7 0.136 -0.012
94 E7 ~~ E12 0.056 -0.009
87 E6 ~~ E10 0.040 0.009
53 E2 ~~ E6 0.012 -0.003
101 E9 ~~ E12 0.011 0.004Die Modifikationsindizes zeigen kovariierende Fehlervariablen von Itempaaren (z.B. zwischen den Items E1 und E4) an. Korrelierte Fehler könnten in einem neuen Testmodell spezifiziert werden. Jedoch sollten wenn möglich nur korrelierte Fehler aufgenommen werden, für die es eine überzeugende Erklärung aus der Theorie gibt. Viele korrelierte Fehler können auch darauf hindeuten, dass den Items eigentlich mehrere latente Variablen zugrunde liegen. Eventuell kann es informativ sein, die Struktur der Items (sowie die optimale Anzahl an latenten Variablen) mit einer Explorativen Faktorenanalyse zu untersuchen.
5. Interpretation der Modellparameter
Die Schätzungen der Modellparameter finden Sie in dem weiter oben präsentierten summary-Output.
Die Spalte \(\texttt{Estimate}\) im Block \(\texttt{Latent Variables}\) zeigt die Schätzwerte für die Steigungsparameter der Items \(\beta_i\). In der Spalte \(\texttt{Std.all}\) finden wir die Schätzwertwerte für die standardisierten Steigungsparameter \(\beta_{zi}\) (Ladungen). Im eindimensionalen \(\tau\)-kongenerischen Modell entspricht die quadrierte standardisierte Ladung dem Anteil der Varianz des Items, der durch die latente Variable erklärt wird (d.h., der Item-Reliabilität).
Beispiel: Das Item E1 hat einen geschätzten Steigungsparameter von \(\hat{\beta}_{E1} = 0.517\) und einen geschätzten standardisierten Steigungsparameter von \(\hat{\beta}_{zE1} = 0.530\).
Interpretation:
- Steigt der Wert auf der latenten Variable Extraversion um eine Einheit, steigt die Itemantwort auf Item E1 im Mittel um \(0.517\) Punkte.
- Steigt der Wert auf der latenten Variable Extraversion um eine Standardabweichung, steigt die Itemantwort auf Item E1 im Mittel um \(0.530\) Standardabweichungen.
- Die latente Variable Extraversion erklärt \(0.53^2 \cdot 100 = 28.09\%\) der Unterschiede in den Itemantworten von Item E1.
Unter \(\texttt{Intercepts}\) finden wir die Schätzwerte für die Schwierigkeitsparameter der Items, die im \(\tau\)-kongenerischen Modell dem Erwartungswert jedes Items entsprechen.
Beispiel: Der geschätzte Schwierigkeitsparameter für Item E2 ist \(\hat{\sigma}_{E2} = 2.723\).
Interpretation:
- Die mittlere Itemantwort auf Item E2 für eine Person mit durchschnittlicher Extraversion beträgt \(2.723\) Punkte.
- Die mittlere Itemantwort auf Item E2 für eine zufällig gezogene Person beträgt \(2.723\) Punkte.
Wenn wir in der Console den Code mean(neo$E2) ausführen, bekommen wir ebenfalls das Ergebnis \(2.723\), da wir mit dem Mittelwert den Erwartungswert \(E(X_{E2})\) schätzen.
Die Spalte \(\texttt{Estimate}\) im Block \(\texttt{Variances}\) zeigt die Schätzwerte für die Fehlervarianzen der Items \(VAR(\varepsilon_i)\). In der Spalte \(\texttt{Std.all}\) finden wir die Schätzwertwerte für die standardisierte Fehlervarianz. Im eindimensionalen \(\tau\)-kongenerischen Modell entspricht die standardisierte Fehlervarianz dem Anteil der Varianz des Items, der nicht durch die latente Variable erklärt wird.
Beispiel: Die geschätzte Fehlervarianz für Item E3 beträgt \(\widehat{VAR}(\varepsilon_{E3}) = 0.560\). Die geschätzte standardisierte Fehlervarianz für Item E3 beträgt \(0.474\).
Interpretation:
- Die latente Variable Extraversion erklärt \((1-0.474) \cdot 100 = 52.6\%\) der Unterschiede in den Itemantworten von Item E3.
Im \(\tau\)-kongenerischen Modell wird durch die Normierung der Erwartungswert der latenten Variable auf 0 fixiert und die Varianz der latenten Variable auf 1.
Die Fixierung der Varianz kann man in unserem Output im Bereich \(\texttt{Variance}\) in der Zeile \(\texttt{e}\) Spalte \(\texttt{Estimate}\) erkennen. Da diese Varianz auf 1 fixiert und nicht geschätzt wurde, wird kein Standardfehler und auch kein Signifikanztest berechnet. In Testmodellen, bei denen die Varianz der latenten Variable frei geschätzt wird, findet sich in dieser Zeile stattdessen der entsprechende Schätzwert.
Wird in einem Testmodell der Erwartungswert der latenten Variable frei geschätzt und nicht fixiert, taucht im Bereich \(\texttt{Intercepts}\) zusätzlich eine Zeile mit dem Namen der latenten Variable (hier \(\texttt{e}\)) auf, in der dann der entsprechende Schätzwert, Standardfehler und Signifikanztest angezeigt wird.
6. Grafische Darstellung des Modells
Mit semPaths() können wir das geschätzte Modell model_fit_extra grafisch darstellen. Die verschiedenen Argumente steuern die Formatierung der Abbildung und werden hier nicht im Detail erläutert. Für andere Datenbeispiele müssen Sie einfach das Modellobjekt austauschen und den Rest des Befehls eins zu eins übernehmen:
semPlot::semPaths(model_fit_extra, whatLabels = "std", intercepts = FALSE,
style = "lisrel", layout = "tree2", rotation = 1)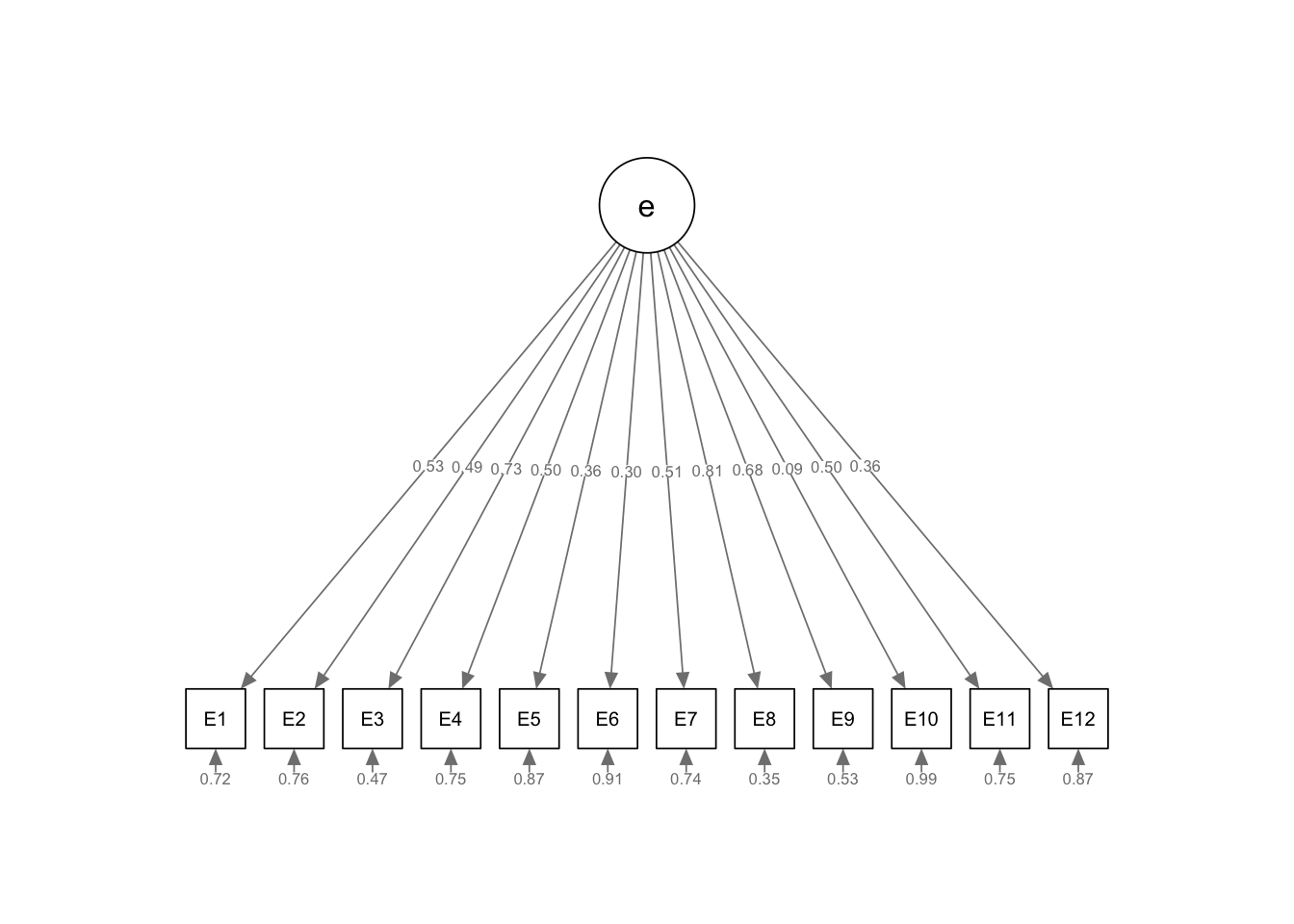
Die latente Variable \(\texttt{e}\) (Extraversion) wird als Kreis dargestellt. Die manifesten Itemantworten \(\texttt{E1,...,E12}\) werden als Rechtecke dargestellt. Die Zahlen neben den kurzen auf die Items weisenden Pfeile geben die Schätzwerte der standardisierten Fehlervarianzen an. Die Zahlen auf den langen Pfeilen zwischen der latenten Variable und den Items geben die Schätzwerte der standardisierten Steigungsparameter an.
3 Arbeitsauftrag
In dem folgenden Arbeitsauftrag können Sie die gleiche Analyse wie oben nochmals mit einem anderen Faktor als Extraversion ausführen. Die Analyse für Ihren Datensatz aus dem UK funktioniert später analog.
Arbeitsauftrag: Führen Sie eine CFA für die Skala Gewissenhaftigkeit (Variablennamen: C1, C2, … , C12) des Beispieldatensatzes NEO_original.csv:
- Schätzen ein eindimensionales \(\tau\)-kongenerische Modell
- Evaluieren Sie die Modellpassung (Modelltest, Fit-Indizes, Signifikanztests für die Parameter, Modifikationsindizes)
- Interpretieren Sie die Schätzwerte der Modellparameter
## CFA:
### Eindimensionales $\tau$-kongenerisches Modell für Gewissenhaftigkeitsitems
model <- "c =~ C1 + C2 + C3 + C4 + C5 + C6 + C7 + C8 + C9 + C10 + C11 + C12"
### Testmodell schätzen
model_fit_gewiss <- cfa(model = model, data = neo, std.lv = TRUE, meanstructure = TRUE)
### Modelltest, Fit-Indices, Informationskriterien, Parameterschätzungen anzeigen
summary(model_fit_gewiss, standardized = TRUE, fit.measures = TRUE)lavaan 0.6.17 ended normally after 20 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 36
Number of observations 566
Model Test User Model:
Test statistic 429.025
Degrees of freedom 54
P-value (Chi-square) 0.000
Model Test Baseline Model:
Test statistic 2174.573
Degrees of freedom 66
P-value 0.000
User Model versus Baseline Model:
Comparative Fit Index (CFI) 0.822
Tucker-Lewis Index (TLI) 0.783
Loglikelihood and Information Criteria:
Loglikelihood user model (H0) -8187.566
Loglikelihood unrestricted model (H1) -7973.054
Akaike (AIC) 16447.132
Bayesian (BIC) 16603.322
Sample-size adjusted Bayesian (SABIC) 16489.039
Root Mean Square Error of Approximation:
RMSEA 0.111
90 Percent confidence interval - lower 0.101
90 Percent confidence interval - upper 0.121
P-value H_0: RMSEA <= 0.050 0.000
P-value H_0: RMSEA >= 0.080 1.000
Standardized Root Mean Square Residual:
SRMR 0.065
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
c =~
C1 0.512 0.039 13.141 0.000 0.512 0.544
C2 0.701 0.043 16.190 0.000 0.701 0.645
C3 0.478 0.043 11.103 0.000 0.478 0.471
C4 0.341 0.026 13.052 0.000 0.341 0.541
C5 0.619 0.037 16.513 0.000 0.619 0.655
C6 0.719 0.048 15.116 0.000 0.719 0.611
C7 0.529 0.034 15.476 0.000 0.529 0.623
C8 0.338 0.027 12.365 0.000 0.338 0.517
C9 0.636 0.044 14.617 0.000 0.636 0.595
C10 0.566 0.031 18.325 0.000 0.566 0.709
C11 0.638 0.042 15.148 0.000 0.638 0.612
C12 0.409 0.043 9.484 0.000 0.409 0.409
Intercepts:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.C1 2.650 0.040 67.033 0.000 2.650 2.818
.C2 2.495 0.046 54.653 0.000 2.495 2.297
.C3 2.611 0.043 61.135 0.000 2.611 2.570
.C4 3.352 0.027 126.457 0.000 3.352 5.315
.C5 2.459 0.040 61.970 0.000 2.459 2.605
.C6 1.989 0.049 40.216 0.000 1.989 1.690
.C7 2.726 0.036 76.338 0.000 2.726 3.209
.C8 3.408 0.028 123.816 0.000 3.408 5.204
.C9 2.555 0.045 56.813 0.000 2.555 2.388
.C10 2.834 0.034 84.493 0.000 2.834 3.551
.C11 2.797 0.044 63.800 0.000 2.797 2.682
.C12 2.558 0.042 60.779 0.000 2.558 2.555
Variances:
Estimate Std.Err z-value P(>|z|) Std.lv Std.all
.C1 0.623 0.040 15.735 0.000 0.623 0.704
.C2 0.688 0.046 14.976 0.000 0.688 0.584
.C3 0.804 0.050 16.089 0.000 0.804 0.778
.C4 0.281 0.018 15.752 0.000 0.281 0.707
.C5 0.509 0.034 14.874 0.000 0.509 0.571
.C6 0.868 0.057 15.282 0.000 0.868 0.627
.C7 0.442 0.029 15.185 0.000 0.442 0.612
.C8 0.314 0.020 15.881 0.000 0.314 0.733
.C9 0.740 0.048 15.408 0.000 0.740 0.646
.C10 0.317 0.022 14.199 0.000 0.317 0.497
.C11 0.680 0.045 15.273 0.000 0.680 0.625
.C12 0.835 0.051 16.306 0.000 0.835 0.833
c 1.000 1.000 1.000## Modifikationsindices anzeigen
modindices(model_fit_gewiss, sort. = TRUE, standardized = FALSE) lhs op rhs mi epc
78 C5 ~~ C7 48.957 0.160
94 C7 ~~ C12 40.915 0.176
95 C8 ~~ C9 40.097 0.139
53 C2 ~~ C6 38.630 0.229
72 C4 ~~ C8 32.041 0.076
104 C11 ~~ C12 26.758 -0.176
54 C2 ~~ C7 24.575 -0.131
48 C1 ~~ C11 24.454 0.148
92 C7 ~~ C10 21.395 0.086
51 C2 ~~ C4 19.644 -0.091
76 C4 ~~ C12 19.119 0.094
44 C1 ~~ C7 19.101 -0.106
93 C7 ~~ C11 19.009 -0.113
79 C5 ~~ C8 17.226 -0.077
59 C2 ~~ C12 16.941 -0.142
58 C2 ~~ C11 15.636 0.129
74 C4 ~~ C10 14.713 0.055
70 C4 ~~ C6 11.778 -0.078
89 C6 ~~ C12 11.502 -0.130
67 C3 ~~ C11 10.807 0.110
66 C3 ~~ C10 10.036 -0.076
57 C2 ~~ C10 9.689 -0.073
39 C1 ~~ C2 9.353 0.093
85 C6 ~~ C8 8.034 -0.068
101 C9 ~~ C12 7.677 -0.098
61 C3 ~~ C5 6.966 0.078
75 C4 ~~ C11 6.330 -0.051
97 C8 ~~ C11 5.828 -0.051
80 C5 ~~ C9 5.783 -0.070
91 C7 ~~ C9 5.467 -0.063
83 C5 ~~ C12 4.900 0.066
81 C5 ~~ C10 4.568 -0.043
103 C10 ~~ C12 4.568 0.052
102 C10 ~~ C11 3.904 -0.045
96 C8 ~~ C10 3.293 0.027
63 C3 ~~ C7 3.215 -0.049
56 C2 ~~ C9 3.069 0.059
50 C2 ~~ C3 3.026 0.059
71 C4 ~~ C7 2.608 0.026
88 C6 ~~ C11 2.531 0.057
69 C4 ~~ C5 2.182 -0.026
55 C2 ~~ C8 2.093 -0.031
84 C6 ~~ C7 1.668 -0.038
42 C1 ~~ C5 1.634 -0.034
46 C1 ~~ C9 1.566 -0.039
77 C5 ~~ C6 1.508 -0.039
41 C1 ~~ C4 1.494 -0.023
65 C3 ~~ C9 1.395 -0.041
47 C1 ~~ C10 1.155 -0.023
60 C3 ~~ C4 1.075 -0.022
43 C1 ~~ C6 1.071 0.035
82 C5 ~~ C11 0.705 0.024
100 C9 ~~ C11 0.623 0.026
64 C3 ~~ C8 0.610 -0.017
86 C6 ~~ C9 0.525 0.027
40 C1 ~~ C3 0.434 0.021
52 C2 ~~ C5 0.333 0.017
73 C4 ~~ C9 0.330 0.012
62 C3 ~~ C6 0.295 -0.021
45 C1 ~~ C8 0.290 -0.011
68 C3 ~~ C12 0.290 0.019
90 C7 ~~ C8 0.254 -0.009
98 C8 ~~ C12 0.140 -0.008
87 C6 ~~ C10 0.121 0.009
99 C9 ~~ C10 0.070 -0.006
49 C1 ~~ C12 0.060 0.008### Geschätztes Modell grafisch darstellen
semPlot::semPaths(model_fit_gewiss, whatLabels = "std", intercepts = FALSE,
style = "lisrel", layout = "tree2", rotation = 1)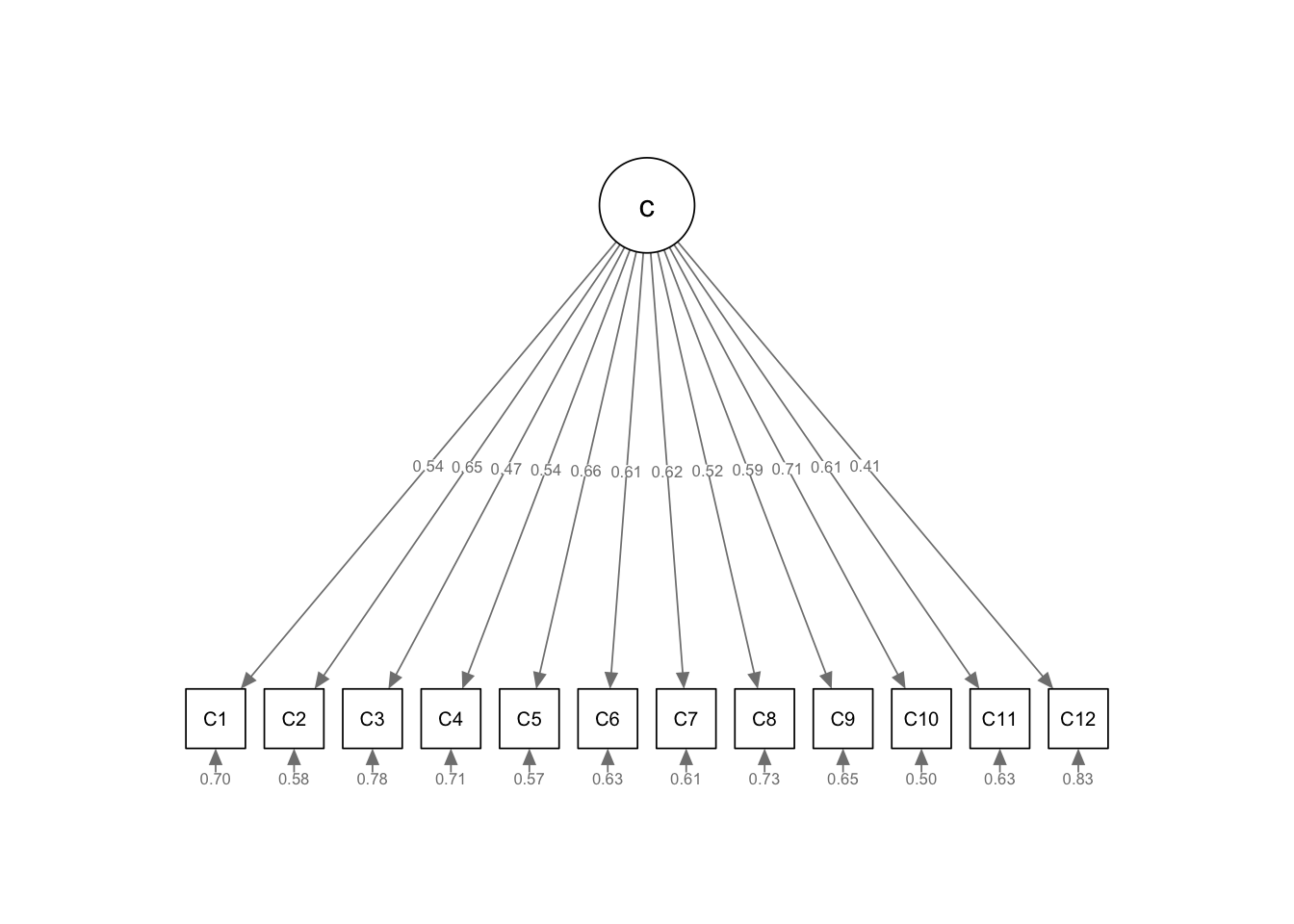
Kurze Zusammenfassung:
- Der Modelltest entscheidet sich für die \(H_1\) und lehnt damit das eindimensionale \(\tau\)-kongenerische Testmodell für die Gewissenhaftigkeitsitems in der Population ab.
- Auch die Fit-Indices RMSEA und CFI zeigen Probleme beim globalen Modellfit an, wohingegen der SRMR basierend auf den groben Richtwerten von Hu & Bentler (1998) ok ausfällt.
- Hinsichtlich des lokalen Modellfits sind die Signifikanztests für die Modellparameter alle signifikant und damit unauffällig.
- Jedoch zeigen die Modifikationsindices korrelierte Messfehler an.
- Für die Interpretation der verschiedenen Modellparameter orientieren Sie sich bitte am Beispiel der Extraversionsskala.
4 Bonus: Strengere Testmodelle
In unserem Datenbeispiel haben wir nur das \(\tau\)-kongenerische Modell betrachtet. Auch bei diesem Testmodell mit den am wenigsten strengen Annahmen, scheint eine deutliche Verletzung des Modellfits vorzuliegen. Daher betrachten wir keine der noch strengeren Modelle. Da ein schlechter Modellfit für das \(\tau\)-kongenerische Modell der Normalfall ist, sind strengere Testmodelle in der Praxis nicht wirklich relevant.
Falls Sie die Analyse der strengeren Testmodelle trotzdem interessiert, können Sie sich diese im Folgenden ansehen.
4.1 \(\tau\)-kongenerisches Modell in einer anderen Schreibweise
Wir nutzen eine andere, ausführlichere Schreibweise für das \(\tau\)-kongenerische Modell:
Das Ergebnis ist dasselbe wie bei dem oberen Befehl
model <- "e =~ NA*E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12
1 E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12 ~ 1
2 e ~ 0
3 e ~~ 1*e"- 1
-
In der 2. Zeile wird festgelegt, dass die Schwierigkeitsparameter frei geschätzt werden sollen:
E1 + ... + E12 ~ 1 - 2
-
\(E(\theta)\) wird auf \(0\) fixiert:
e ~ 0 - 3
-
\(Var(\theta)\) wird auf \(1\) fixiert:
e ~~ 1*e
model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)lavaan 0.6.17 ended normally after 16 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 36
Number of observations 566
Model Test User Model:
Test statistic 495.835
Degrees of freedom 54
P-value (Chi-square) 0.000
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
e =~
E1 0.517 0.041 12.636 0.000
E2 0.418 0.036 11.668 0.000
E3 0.788 0.042 18.721 0.000
E4 0.425 0.036 11.951 0.000
E5 0.378 0.046 8.218 0.000
E6 0.306 0.046 6.689 0.000
E7 0.502 0.042 12.039 0.000
E8 0.710 0.033 21.682 0.000
E9 0.751 0.043 17.282 0.000
E10 0.094 0.048 1.955 0.051
E11 0.471 0.040 11.860 0.000
E12 0.400 0.048 8.308 0.000
Intercepts:
Estimate Std.Err z-value P(>|z|)
.E1 2.214 0.041 53.964 0.000
.E2 2.723 0.036 76.530 0.000
.E3 2.473 0.046 54.123 0.000
.E4 2.928 0.035 82.641 0.000
.E5 1.809 0.044 40.947 0.000
.E6 1.776 0.043 40.876 0.000
.E7 1.760 0.042 42.342 0.000
.E8 2.673 0.037 72.250 0.000
.E9 2.337 0.046 50.566 0.000
.E10 1.963 0.045 43.909 0.000
.E11 2.389 0.039 60.555 0.000
.E12 1.890 0.046 40.876 0.000
e 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
e 1.000
.E1 0.685 0.044 15.717 0.000
.E2 0.541 0.034 15.907 0.000
.E3 0.560 0.041 13.621 0.000
.E4 0.529 0.033 15.854 0.000
.E5 0.962 0.059 16.402 0.000
.E6 0.974 0.059 16.551 0.000
.E7 0.725 0.046 15.837 0.000
.E8 0.271 0.024 11.488 0.000
.E9 0.646 0.045 14.317 0.000
.E10 1.122 0.067 16.800 0.000
.E11 0.659 0.042 15.871 0.000
.E12 1.050 0.064 16.392 0.000Aufgrund der ausführlicheren Modell-Schreibweise müssen die Argumente std.lv = TRUE und meanstructure = TRUE diesmal nicht im Modelltest gesetzt werden.
4.2 essentiell \(\tau\)-äquivalentes Modell für Extraversion:
1model <- "e =~ 1*E1 + 1*E2 + 1*E3 + 1*E4 + 1*E5 + 1*E6 +
1*E7 + 1*E8 + 1*E9 + 1*E10 + 1*E11 + 1*E12
E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12 ~ 1
2 e ~ 0
3 e ~~ e"- 1
-
Steigungsparameter \(\beta\) auf \(1\) fixieren:
~ 1*E1 + ... 1*E12 - 2
-
\(E(\theta)\) auf \(0\) fixieren:
e ~ 0 - 3
- Die Varianz wird frei geschätzt (und nicht wie im \(\tau\)-kongenerischen Modell auf \(1\) fixiert)
model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)lavaan 0.6.17 ended normally after 20 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 25
Number of observations 566
Model Test User Model:
Test statistic 687.208
Degrees of freedom 65
P-value (Chi-square) 0.000
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
e =~
E1 1.000
E2 1.000
E3 1.000
E4 1.000
E5 1.000
E6 1.000
E7 1.000
E8 1.000
E9 1.000
E10 1.000
E11 1.000
E12 1.000
Intercepts:
Estimate Std.Err z-value P(>|z|)
.E1 2.214 0.039 56.550 0.000
.E2 2.723 0.038 70.841 0.000
.E3 2.473 0.042 58.298 0.000
.E4 2.928 0.036 81.235 0.000
.E5 1.809 0.045 40.262 0.000
.E6 1.776 0.046 38.571 0.000
.E7 1.760 0.042 42.280 0.000
.E8 2.673 0.035 77.148 0.000
.E9 2.337 0.043 53.763 0.000
.E10 1.963 0.050 39.079 0.000
.E11 2.389 0.040 60.159 0.000
.E12 1.890 0.047 40.288 0.000
e 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
e 0.263 0.019 13.801 0.000
.E1 0.605 0.039 15.513 0.000
.E2 0.573 0.037 15.441 0.000
.E3 0.756 0.048 15.776 0.000
.E4 0.472 0.031 15.145 0.000
.E5 0.880 0.055 15.924 0.000
.E6 0.937 0.059 15.979 0.000
.E7 0.718 0.046 15.720 0.000
.E8 0.417 0.028 14.920 0.000
.E9 0.807 0.051 15.843 0.000
.E10 1.165 0.072 16.145 0.000
.E11 0.630 0.040 15.565 0.000
.E12 0.983 0.061 16.019 0.0004.3 \(\tau\)-äquivalentes Modell für Extraversion:
model <- "e =~ 1*E1 + 1*E2 + 1*E3 + 1*E4 + 1*E5 + 1*E6 +
1*E7 + 1*E8 + 1*E9 + 1*E10 + 1*E11 + 1*E12
1 E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12 ~ 0
2 e ~ 1
e ~~ e"- 1
-
Dieses Modell hat keine Schwierigkeitsparameter, deshalb steht hier
~ 0 - 2
-
wir brauchen also auch keine Normierung für den Erwartungswert:
e ~ 1
model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)lavaan 0.6.17 ended normally after 32 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 14
Number of observations 566
Model Test User Model:
Test statistic 1980.764
Degrees of freedom 76
P-value (Chi-square) 0.000
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
e =~
E1 1.000
E2 1.000
E3 1.000
E4 1.000
E5 1.000
E6 1.000
E7 1.000
E8 1.000
E9 1.000
E10 1.000
E11 1.000
E12 1.000
Intercepts:
Estimate Std.Err z-value P(>|z|)
.E1 0.000
.E2 0.000
.E3 0.000
.E4 0.000
.E5 0.000
.E6 0.000
.E7 0.000
.E8 0.000
.E9 0.000
.E10 0.000
.E11 0.000
.E12 0.000
e 2.316 0.024 96.159 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
e 0.258 0.020 13.188 0.000
.E1 0.624 0.041 15.322 0.000
.E2 0.755 0.048 15.585 0.000
.E3 0.787 0.050 15.635 0.000
.E4 0.888 0.056 15.771 0.000
.E5 1.151 0.072 16.012 0.000
.E6 1.247 0.078 16.074 0.000
.E7 1.046 0.066 15.930 0.000
.E8 0.559 0.037 15.148 0.000
.E9 0.804 0.051 15.660 0.000
.E10 1.282 0.080 16.095 0.000
.E11 0.626 0.041 15.329 0.000
.E12 1.177 0.073 16.030 0.0004.4 essentiell paralleles Modell für Extraversion:
model <- "e =~ 1*E1 + 1*E2 + 1*E3 + 1*E4 + 1*E5 + 1*E6 +
1*E7 + 1*E8 + 1*E9 + 1*E10 + 1*E11 + 1*E12
1 E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12 ~ 1
2 E1 ~~ var*E1
E2 ~~ var*E2
E3 ~~ var*E3
E4 ~~ var*E4
E5 ~~ var*E5
E6 ~~ var*E6
E7 ~~ var*E7
E8 ~~ var*E8
E9 ~~ var*E9
E10 ~~ var*E10
E11 ~~ var*E11
E12 ~~ var*E12
3 e ~ 0
4 e ~~ e"- 1
-
Wir haben wieder Schwierigkeitsparameter, deshalb steht hier
~ 1 - 2
-
Die Varianzen sollen gleich sein:
E1 ~~ var*E1etc. - 3
-
Normierung:
e ~ 0 - 4
-
Varianz wird frei geschätzt:
e ~~ e
model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)lavaan 0.6.17 ended normally after 23 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 25
Number of equality constraints 11
Number of observations 566
Model Test User Model:
Test statistic 922.984
Degrees of freedom 76
P-value (Chi-square) 0.000
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
e =~
E1 1.000
E2 1.000
E3 1.000
E4 1.000
E5 1.000
E6 1.000
E7 1.000
E8 1.000
E9 1.000
E10 1.000
E11 1.000
E12 1.000
Intercepts:
Estimate Std.Err z-value P(>|z|)
.E1 2.214 0.042 52.847 0.000
.E2 2.723 0.042 64.994 0.000
.E3 2.473 0.042 59.047 0.000
.E4 2.928 0.042 69.886 0.000
.E5 1.809 0.042 43.189 0.000
.E6 1.776 0.042 42.387 0.000
.E7 1.760 0.042 42.008 0.000
.E8 2.673 0.042 63.813 0.000
.E9 2.337 0.042 55.799 0.000
.E10 1.963 0.042 46.858 0.000
.E11 2.389 0.042 57.023 0.000
.E12 1.890 0.042 45.129 0.000
e 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
.E1 (var) 0.748 0.013 55.794 0.000
.E2 (var) 0.748 0.013 55.794 0.000
.E3 (var) 0.748 0.013 55.794 0.000
.E4 (var) 0.748 0.013 55.794 0.000
.E5 (var) 0.748 0.013 55.794 0.000
.E6 (var) 0.748 0.013 55.794 0.000
.E7 (var) 0.748 0.013 55.794 0.000
.E8 (var) 0.748 0.013 55.794 0.000
.E9 (var) 0.748 0.013 55.794 0.000
.E10 (var) 0.748 0.013 55.794 0.000
.E11 (var) 0.748 0.013 55.794 0.000
.E12 (var) 0.748 0.013 55.794 0.000
e 0.245 0.018 13.390 0.0004.5 paralleles Modell für Extraversion:
model <- "e =~ 1*E1 + 1*E2 + 1*E3 + 1*E4 + 1*E5 + 1*E6 +
1*E7 + 1*E8 + 1*E9 + 1*E10 + 1*E11 + 1*E12
1 E1 + E2 + E3 + E4 + E5 + E6 + E7 + E8 + E9 + E10 + E11 + E12 ~ 0
E1 ~~ var*E1
E2 ~~ var*E2
E3 ~~ var*E3
E4 ~~ var*E4
E5 ~~ var*E5
E6 ~~ var*E6
E7 ~~ var*E7
E8 ~~ var*E8
E9 ~~ var*E9
E10 ~~ var*E10
E11 ~~ var*E11
E12 ~~ var*E12
2 e ~ 1
3 e ~~ e"- 1
-
Da wir keine Schwierigkeitsparameter haben steht in der 2. Zeile:
~ 0 - 2
-
Der Erwartungswert der latenten Variable wird frei geschätzt:
e ~ 1 - 3
-
Die Varianz der latenten Variable wird frei geschätzt:
e ~~ e
model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)model_fit_extra <- cfa(model = model, data = neo)
summary(model_fit_extra)lavaan 0.6.17 ended normally after 23 iterations
Estimator ML
Optimization method NLMINB
Number of model parameters 14
Number of equality constraints 11
Number of observations 566
Model Test User Model:
Test statistic 2161.582
Degrees of freedom 87
P-value (Chi-square) 0.000
Parameter Estimates:
Standard errors Standard
Information Expected
Information saturated (h1) model Structured
Latent Variables:
Estimate Std.Err z-value P(>|z|)
e =~
E1 1.000
E2 1.000
E3 1.000
E4 1.000
E5 1.000
E6 1.000
E7 1.000
E8 1.000
E9 1.000
E10 1.000
E11 1.000
E12 1.000
Intercepts:
Estimate Std.Err z-value P(>|z|)
.E1 0.000
.E2 0.000
.E3 0.000
.E4 0.000
.E5 0.000
.E6 0.000
.E7 0.000
.E8 0.000
.E9 0.000
.E10 0.000
.E11 0.000
.E12 0.000
e 2.245 0.023 96.271 0.000
Variances:
Estimate Std.Err z-value P(>|z|)
.E1 (var) 0.912 0.016 55.794 0.000
.E2 (var) 0.912 0.016 55.794 0.000
.E3 (var) 0.912 0.016 55.794 0.000
.E4 (var) 0.912 0.016 55.794 0.000
.E5 (var) 0.912 0.016 55.794 0.000
.E6 (var) 0.912 0.016 55.794 0.000
.E7 (var) 0.912 0.016 55.794 0.000
.E8 (var) 0.912 0.016 55.794 0.000
.E9 (var) 0.912 0.016 55.794 0.000
.E10 (var) 0.912 0.016 55.794 0.000
.E11 (var) 0.912 0.016 55.794 0.000
.E12 (var) 0.912 0.016 55.794 0.000
e 0.232 0.018 12.630 0.000